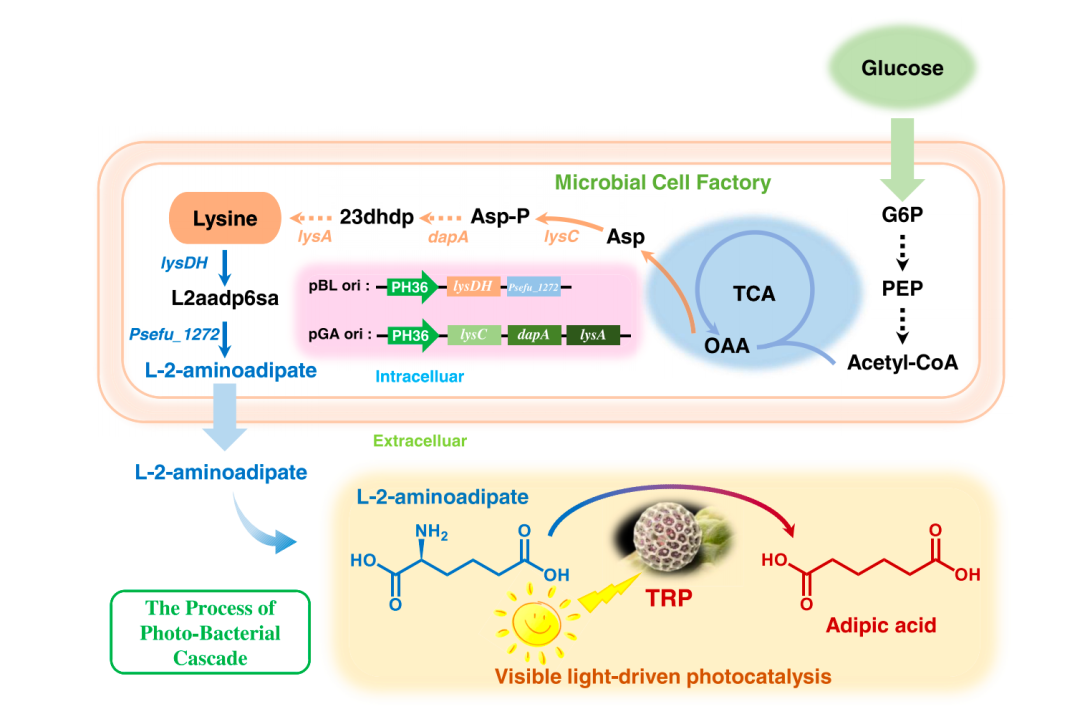
近年来,抗生素耐药问题日益严重,传统抗菌药物的效果正在逐渐减弱,全球科学界正努力寻找全新的抗菌分子来源。
动物毒液是一类高度复杂的天然产物,包含大量具有生物活性的蛋白质和肽类分子,被认为是潜在的“天然药库”。不过,毒液中结构多样的蛋白往往很难直接用于开发药物。
针对这一挑战,近日,美国宾夕法尼亚大学 Cesar de la Fuente-Nunez 教授团队在 Nature Communications 期刊发表了一篇题为“Decoding encrypted antimicrobial peptide signals from venom proteins using deep learning”的研究,研究团队首次将人工智能用于系统分析毒液蛋白质序列,并提出了一种基于深度学习的毒液蛋白组挖掘策略,成功从蜘蛛、蛇等多种生物的毒液中识别出数百种具有抗菌潜力的“隐藏肽段”,为开发新一代抗菌肽提供了全新的研究路径。
该研究的核心思想是通过深度学习技术,在海量毒液蛋白中挖掘出可能具备抗菌活性的短链肽片段。研究团队首先从 4 个数据库(包括蜘蛛、蛇类等 6 类生物)中收集了 16,000 多条毒液蛋白序列。
随后,研究人员通过程序将这些蛋白“切片”,生成总计超过 4000 万个短肽段(每段约 8–50 个氨基酸),并将这些片段输入到一个已训练好的 AI 模型中,用于预测其是否具备抗菌能力。AI 模型的判断依据包括肽段的电荷、疏水性、结构特征等。最终,系统筛选出了 386 种在结构和功能上与已知抗菌肽不同的候选药物。它们表现出高净电荷和高疏水性,这些特性有利于破坏细菌膜。
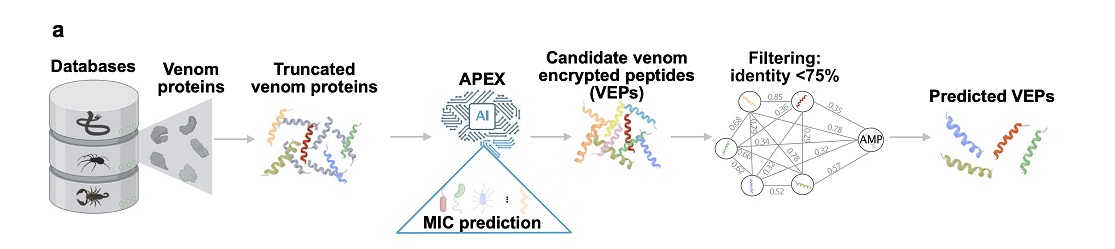 图|毒液蛋白经切片、筛选,得到具有抗菌潜力的短肽片段
图|毒液蛋白经切片、筛选,得到具有抗菌潜力的短肽片段
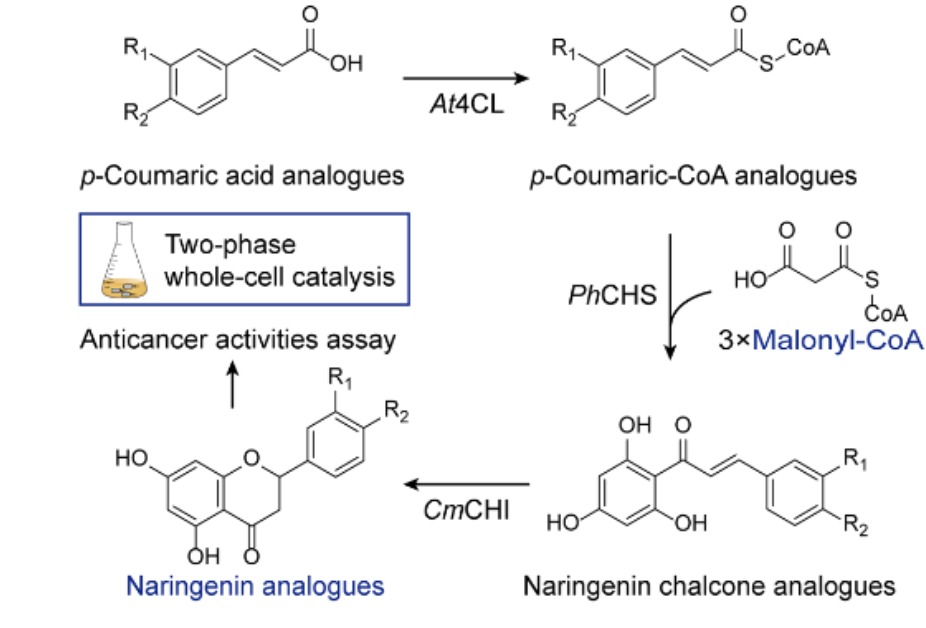
为了验证这些 AI 挑选出的肽段是否真的有效,研究团队在实验室中合成了部分候选肽,并在常见细菌模型中进行测试。
结果显示,多数肽段对金黄色葡萄球菌、大肠杆菌等细菌有明显抑制作用,部分肽段的效果甚至不逊于现有抗生素。进一步分析表明,这些毒液肽(VEPs)大多通过破坏细菌细胞膜发挥杀菌作用,展现出典型的 “膜靶向机制”。这种作用方式对抗耐药菌株至关重要——由于其不依赖细菌的传统代谢通路,能绕过耐药菌通过基因变异形成的常规防御体系,从而有效应对细菌耐药性难题。
与传统天然抗菌肽相比,这些毒液来源的 VEPs 在结构上表现出更多样化,例如部分肽段带有较高的阳离子电荷密度或独特的疏水区域,有助于提高它们对细菌膜的识别和附着能力。
此外,由于毒液肽通常源于动物防御系统,具备较强的稳定性和生物活性,这使得它们更容易被进一步开发为抗菌药物。研究团队还尝试对部分 VEPs 进行修饰,如引入 D-氨基酸或构建环形结构,以提升其在血液中的稳定性和体内半衰期。结果显示,这些修饰后的 VEPs 不仅抗菌效果更强,还对哺乳动物细胞的毒性更低,安全性更高。
除了直接杀菌外,该团队还探讨了 VEPs 的组合使用方式,他们通过拼接多个不同的肽段或构建多肽复合物,进一步提高了其广谱抗菌能力。在小鼠感染模型中,部分组合型 VEPs 能显著清除体内的耐药菌感染,表现出良好的治疗潜力。这一结果提示,毒液中不仅蕴含大量潜在抗菌分子,而且通过合理设计与组合,可以构建出一系列新型的多功能抗菌肽,为临床用药带来更多可能。
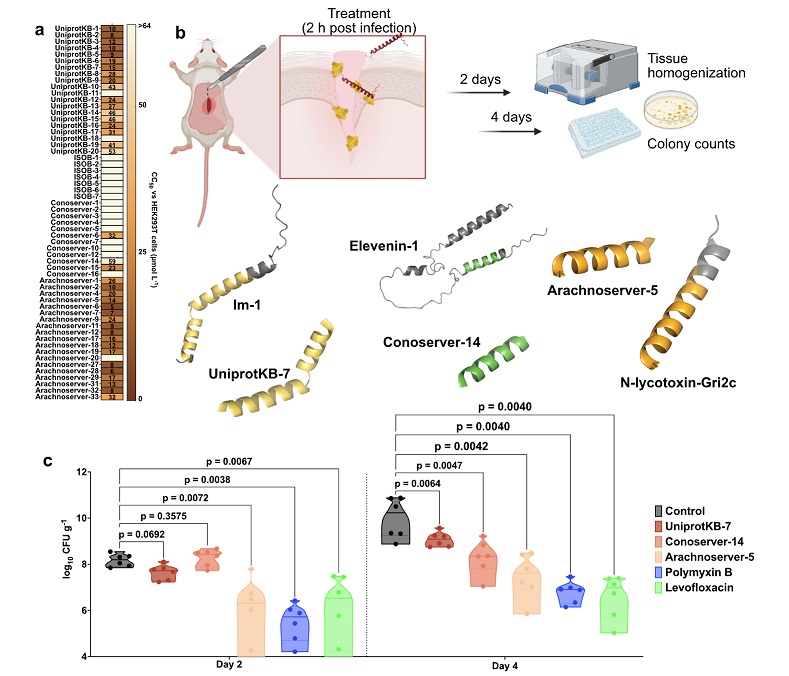
图|小鼠模型实验显示,部分组合 VEPs 在体内抗感染效果显著优于常规药物
总之,该研究首次利用深度学习手段系统性地分析毒液蛋白组,在海量序列中“解码”出隐藏的抗菌信号,建立了一个高效、可扩展的抗菌肽挖掘平台。与传统通过实验逐一筛选的方法相比,AI 驱动的策略不仅速度更快、命中率更高,还能发现过去被忽视的潜在功能片段。该方法不仅适用于蛇毒,也可拓展至其他天然蛋白来源,如海洋生物、昆虫毒素等,为未来解决抗菌耐药问题带来了新思路,也展示了人工智能在天然产物药物开发中的巨大潜力。
参考文献:
1. Guan, C., Torres, M.D.T., Li, S. et al. Computational exploration of global venoms for antimicrobial discovery with Venomics artificial intelligence. Nat Commun 16, 6446 (2025). https://doi.org/10.1038/s41467-025-60051-6
免责声明:本文旨在传递合成生物学最新讯息,不代表平台立场,不构成任何投资意见和建议,以官方/公司公告为准。本文也不是治疗方案推荐,如需获得治疗方案指导,请前往正规医院就诊。


























































































安各洛(深圳)生物科技有限公司 版权所有